单细胞转录组测序
产品名称: 单细胞转录组测序
英文名称: Single -cell RNA -sequencing
产品编号:
产品价格: ¥36000-40000(视平台及样本数决定)
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 :
- 地址 : 上海市浦东新区张江高科技园区爱迪生路332号
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 点击查看
- 传真 : 点击查看
- 邮箱 : service@genechem.com.cn
单细胞测序
单细胞转录组测序( Single -cell RNA -sequencing)是指在单细胞水平上对 RNA 进行高通量测序和分析的新技术。 不同于常规组织或细胞群测序得到的结果(只是大量细胞平均表达水平),单测序能够深入挖掘特异性的信息。 目前 ,单细胞测序已广泛应用于肿瘤异质性、免疫微环境神经科异质性、免疫微环境神经科 、胚胎发育细胞分化等领域的研究。
单细胞测序的原理和平台:
BD Rhapsody(基于microwell的原理)
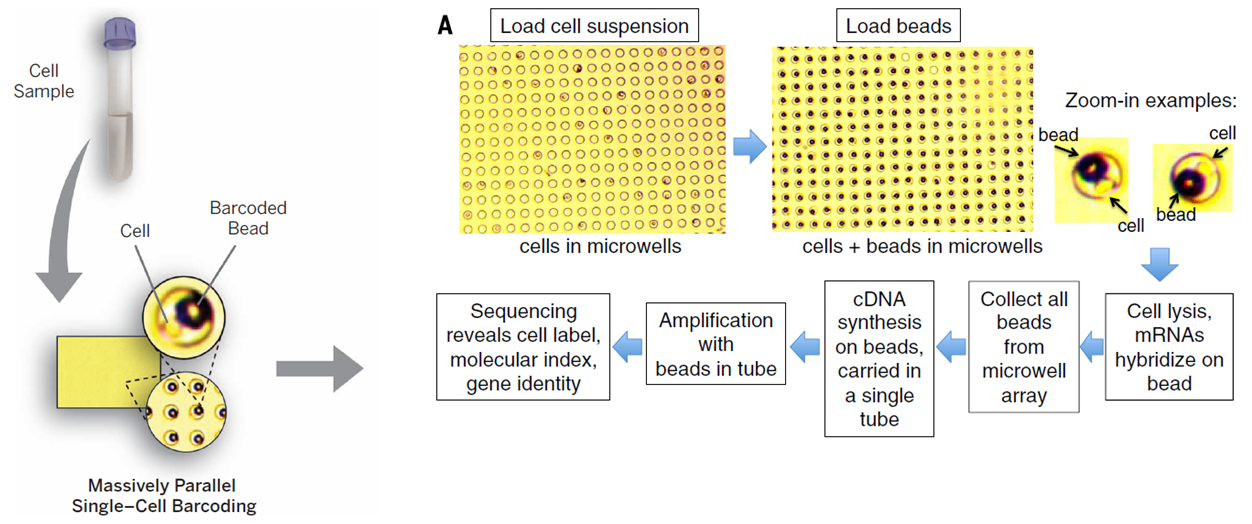
(来源于文献)
10X genomics (基于DROP-seq的原理)
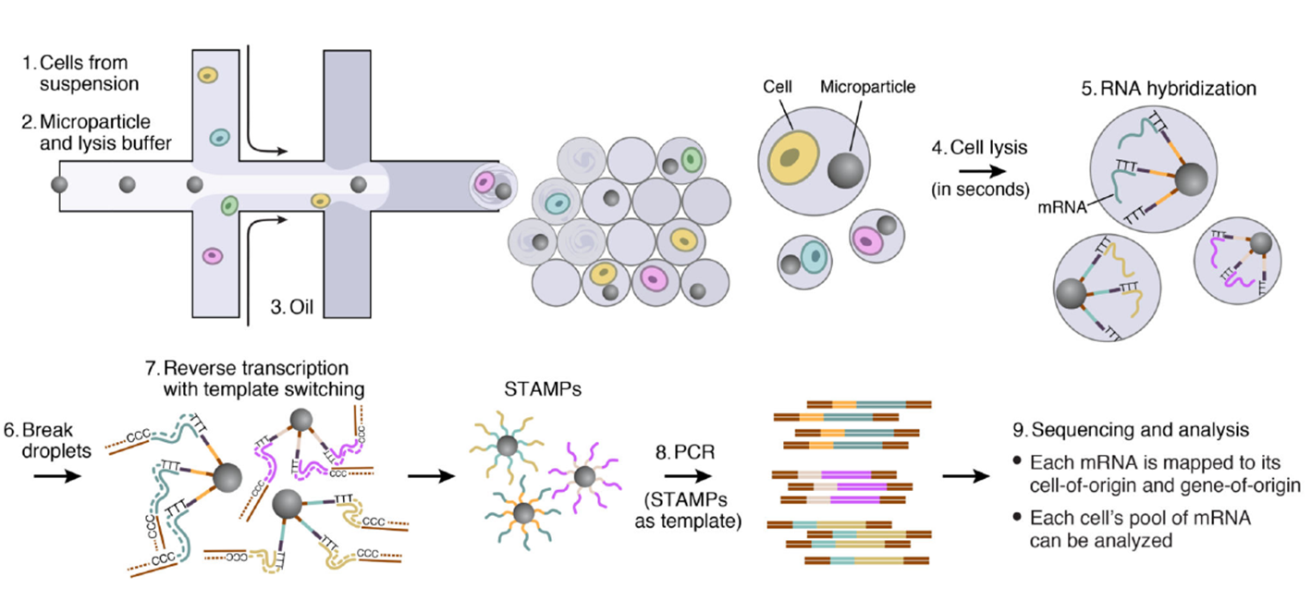
(来源于文献)
技术优势
对于多细胞生物来说,细胞与细胞之间是存在差异的。受精卵从一个细胞开始分裂到最终发育成成熟的个体,各种功能细胞之间的差异会越来越大,表达各自不同的基因,决定不同的生理功能。疾病的发生过程中,伴随多种细胞的参与,不同细胞也会受到不同影响,如肿瘤的异质性、原发灶、转移灶、肿瘤微环境细胞等其基因表达有很大的差异,这些差异决定了肿瘤细胞对治疗的反应。
传统的研究方法由于技术限制,是在多细胞水平进行的,获得的基因表达信息是细胞群体的平均信号,丢失了细胞个体特异的基因表达信息。为了更加精细地研究基因表达与细胞功能的关系,单细胞水平进行检测是更加准确的方法,获得的基因表达信息也更加直观。单细胞测序的发展将基因表达与细胞功能的研究带入了一个新的时代。
单细胞测序解决了单个细胞RNA量少的问题:
在单细胞测序出现之前,主要是Bulk RNA-seq,即测量一个大的细胞群体中每一个基因的平均表达水平,但对于基因表达的本质研究不够深入,很多基因表达的时空信息被掩盖。scRNA-seq直到2014年后才降测序费用降低到可接受的水平,逐渐进入大家的视野。它测定的是细胞种群中每个细胞中的基因的表达量分布,对于研究特定细胞基因表达的变化意义重大。能同时测定的细胞数量从最初的几十上百个到现在的成千上万个,向着高通量的方向快速发展。将研究推升到一个更加精准的方向。
单细胞测序可以解决的科学问题
1.研究对象中细胞一共分多少群?
2.每个群对应哪种细胞?
3.如何定义新的细胞类群?
4.已知的某类细胞是否可分成几个亚群?
5.每个细胞群/亚群的marker基因?
6.不同样本(病人)间的细胞构成有何差异?
7.细胞构成和基因表达差异与表型的关系?
技术参数
样本的要求
1. 总量> 104目标细胞个数(最少10,000个细胞);
2. 浓度500-1,000个/uL;
3. 细胞间无粘连(成团率<5%);
4. 无大于40um的细胞碎片或其他颗粒物;
5. 细胞成活率应大于80%(台盼蓝染色检测);
6. 不存在逆转录抑制剂和非细胞的核酸分子。
常规项目周期
45-50个工作日
分析内容
| 单细胞转录组 | 分析内容 |
| 标准分析 | 1.数据预处理 |
| 1.1基因组比对及基因表达定量 | |
| 1.2基因及样本过滤 | |
| 1.3细胞周期效应评估 | |
| 2.细胞聚类和可视化 | |
| 2.1 louvain算法聚类 | |
| 2.2 T-SNE、umap、Force-directed graph drawing可视化 | |
| 高级分析 | 3.细胞类型注释 |
| 3.1细胞类型注释及可视化 | |
| 3.2 Marker基因heatmap | |
| 3.3 Marker基因小提琴图 | |
| 4. Marker基因分析 | |
| 4.1 Logistic Regression算法 | |
| 4.2 t-test_overestim算法 | |
| 4.3 Wilcoxon算法 | |
| 5.多样本比较分析 | |
| 5.1各样本中的细胞类型分布比例统计 | |
| 5.2各样本和Cluster中细胞数目统计 | |
| 5.3各样本和Cluster中转录本表达丰度统计 | |
| 5.4各样本和cluster中基因数目统计 |
细胞群体能被分成多种亚群
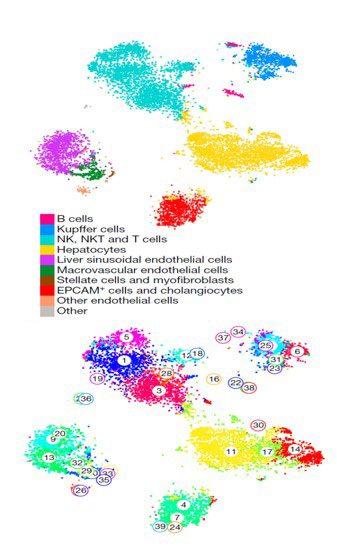
不同亚群的细胞表达不同的marker基因
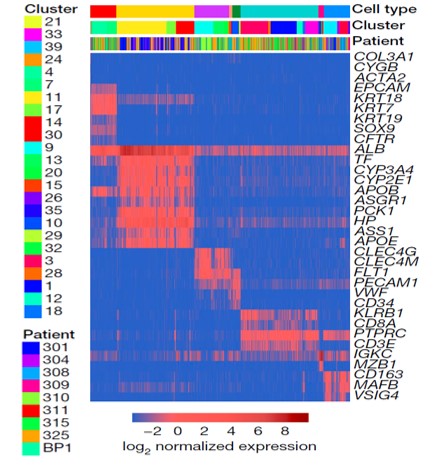
相同的基因在不同的细胞中呈现差异表达,细胞的功能和命运也因此而不同
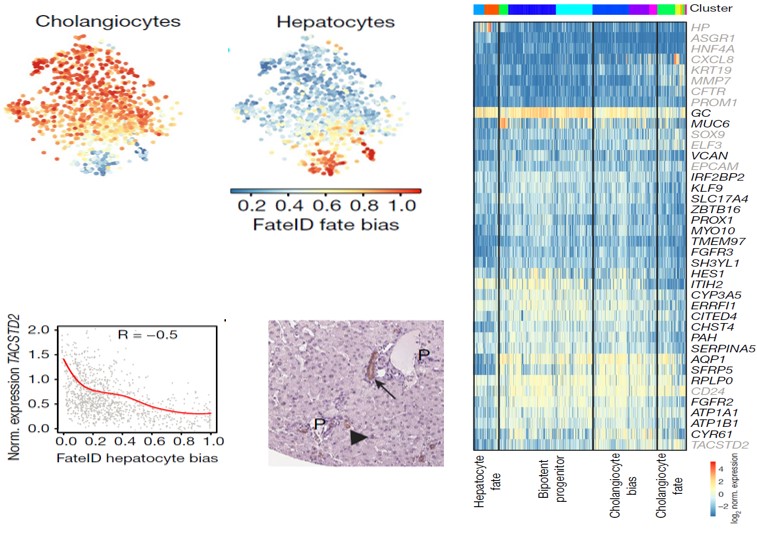
参考文献
1.Aizarani, N. et al. A human liver cell atlas reveals heterogeneity and epithelial progenitors. Nature 572, 199-204, doi:10.1038/s41586-019-1373-2 (2019).
2.Birey, F. et al. Assembly of functionally integrated human forebrain spheroids. Nature 545, 54-59, doi:10.1038/nature22330 (2017).
3.Fan, H. C., Fu, G. K. & Fodor, S. P. Expression profiling. Combinatorial labeling of single cells for gene expression cytometry. Science 347, 1258367, doi:10.1126/science.1258367 (2015).
4.Azizi, E. et al. Single-Cell Map of Diverse Immune Phenotypes in the Breast Tumor Microenvironment. Cell 174, 1293-1308 e1236, doi:10.1016/j.cell.2018.05.060 (2018).
5.Zheng, C. et al. Landscape of Infiltrating T Cells in Liver Cancer Revealed by Single-Cell Sequencing. Cell 169, 1342-1356 e1316, doi:10.1016/j.cell.2017.05.035 (2017).
6.Young, M. D. et al. Single-cell transcriptomes from human kidneys reveal the cellular identity of renal tumors. Science 361, 594-599, doi:10.1126/science.aat1699 (2018).
