BioNano单分子光学图谱
产品名称: BioNano单分子光学图谱
英文名称: BioNano
产品编号:
产品价格: 询价
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 :
- 地址 : 上海市松江区九亭镇中心路1158号5号楼5层
- 邮编 : 200233
- 所在区域 : 上海
- 电话 : 400***0612 点击查看
- 传真 : 点击查看
- 邮箱 : market2@genenergy.cn
BioNano单分子光学图谱:
NGS可以获得大量的测序数据但是由于读长较短,无法直观的看到整个基因组范围的结构变化,而且由于基因组的复杂性(重复序列和结构变异)限制了基因组的重构和对功能原件的理解。因此迫切需要针对更长片段的分析方法。BioNano公司利用其专利的芯片技术开发的Irys系统可以对长链DNA单分子成像,在更短时间提供更完整的基因图谱
Irys系统特点:
l 长链DNA分子 ---可检测长达几百kb的长链DNA分子,很好解决重复序列和可变区对研究带来的影响。
l 高质量的数据 ---单分子DNA通过纳米通道被拉直平行排列,利用高质量图像使基因组结构展现无遗。
l 应用灵活 ---开放性的平台适合研究人员进行多个领域的研究,尤其在de novo和染色体结构变异方面提供了全新的研究方法。
技术路线

生物信息学分析模块
|
BIONANO-01 |
测序质量评估(QC)与预处理 |
|
BIONANO-02 |
Denovo拼接分析(无参考基因组) |
|
BIONANO-03 |
结构变异分析(有参考基因组) |
部分结果展示
|
=== Molecule Size Distribution === |
||||
|
Length Bin |
# of Molecules |
Quantity (kb) |
Bin Proportion (% by mass) |
Labels (/100kb) |
|
10-500kb |
3,454 |
903,099 |
83% |
13.29 |
|
150-500kb |
3,454 |
903,099 |
83% |
13.29 |
|
200-500kb |
2,466 |
730,422 |
67% |
13.34 |
|
250-500kb |
1,609 |
537,868 |
49% |
13.41 |
|
500-5000kb |
301 |
190,668 |
17% |
14.27 |
单分子长度分布统计
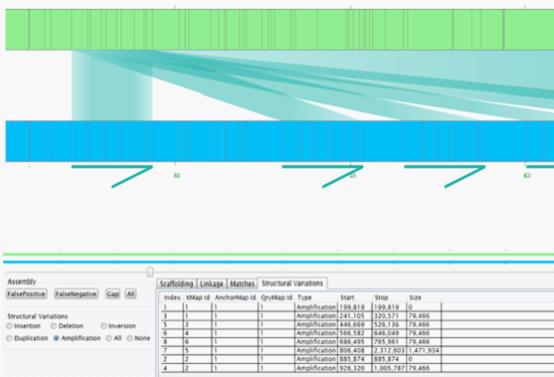
结构变异结果
|
|
---- Assembly Report --------- |
|
Molecule Size Cutoff |
150kb |
|
Label Number Cutoff |
10 |
|
Input Quantity (Mb) |
709.3 |
|
Input Molecule N50 (kb) |
291 |
|
Avg Label Density (per 100 kb) |
12.2 |
|
Number of Consensus Genome Maps |
2 |
|
Total Size of Consensus Genome Maps (Mb) |
4.8 |
|
N50 Consensus Genome Maps (Mb) |
3.9 |
|
Avg Depth of Coverage |
107.1 |
De novo拼接统计
样品要求
A:植物样品
建议以植物种子萌发幼苗来提取。由种子萌发的1~2叶期的嫩苗,在采集前1~3天,避光黄化处理。嫩苗用液氮速冻后,保存-80度或干冰中运输。一次DNA提取需要~5g植物样品,建议提供给我们的样品量为15~20g;无法提供种子的样品,可以采集相应量的茎尖组织。
B:昆虫
5g,虫卵最佳,其次幼虫,成虫难提取。
C:血液
新鲜血液10ml,EDTA抗凝。采集鲜血后5日内有效,冰箱存放,冰盒运输,不用冰袋,以防局部温度过低。避免使用有溶血或凝血的血液。
参考文献
[1] Lam ET, Hastie A, Lin C, Ehrlich D, Das SK, Austin MD, Deshpande P, Cao H, Nagarajan N, Xiao M, Kwok PY. Genome mapping on nanochannel arrays for structural variation analysis and sequence assembly. Nat Biotechnol. 2012 Aug;30(8):771-6.
[2] Das SK, Austin MD, Akana MC, Deshpande P, Cao H, Xiao M. Single molecule linear analysis of DNA in nano-channel labeled with sequence specific fluorescent probes. Nucleic Acids Res. 2010 Oct;38(18):e177. doi: 10.1093/nar/gkq673. Epub 2010 Aug 10
应用案例
PLOS ONE. 2013;8(2):e55864. doi: 10.1371.
Rapid Genome Mapping in Nanochannel Arrays for Highly Complete and Accurate De Novo Sequence Assembly of the Complex Aegilops tauschii Genome.
Hastie AR, Dong L, Smith A, Finklestein J, Lam ET, Huo N, Cao H, Kwok PY, Deal KR, Dvorak J, Luo MC, Gu Y, Xiao M.
背景:
TUNEology 波长可调检测卡盒-->
