
1.核酸测序
高通量测序技术(NextGen)
微生物的前沿研究主要依托于核酸测序速度和通量的增加。在1977-2005年间,主要的测序方法为Sanger双脱氧链终止法,但是如果利用它进行单个细菌基因组测序可能需要花费多年的时间,因此随着高通量测序技术的推出,Sanger法很快就退出了主流舞台。
高通量测序技术使用了多个高通量测序平台:第一种是Roche GS20 454(罗氏的454平台,2005-2016),该平台主要基于焦磷酸测序原理。虽然454测序是一个关键的技术进步,但是该平台存在测序试剂昂贵、复杂重复序列错误率高以及磁珠载荷存在局限等缺陷,从而限制了其通量和获得的序列条数。第二种是Solexa(现在的Illumina)Genome Analyzer (GA),该平台借助于边合成边测序、可逆阻断和桥式PCR等技术,被广泛应用于人、微生物等领域的核酸测序研究,而且个别仪器单个run可产生>1.8 TB的数据。Illumina目前包括GA, MiSeq 和HiSeq系列等多个测序平台,可以进行100-300 bp 双端测序,有望实现每年进行大于18000个人的基因组测序(3 Gbp,每个人1000美元)。此外,Illumina还推出了TruSeq(Moleculo)平台,读长可达到>8 kbp ,该平台推进了高复杂微生物群落中微生物序列的组装。
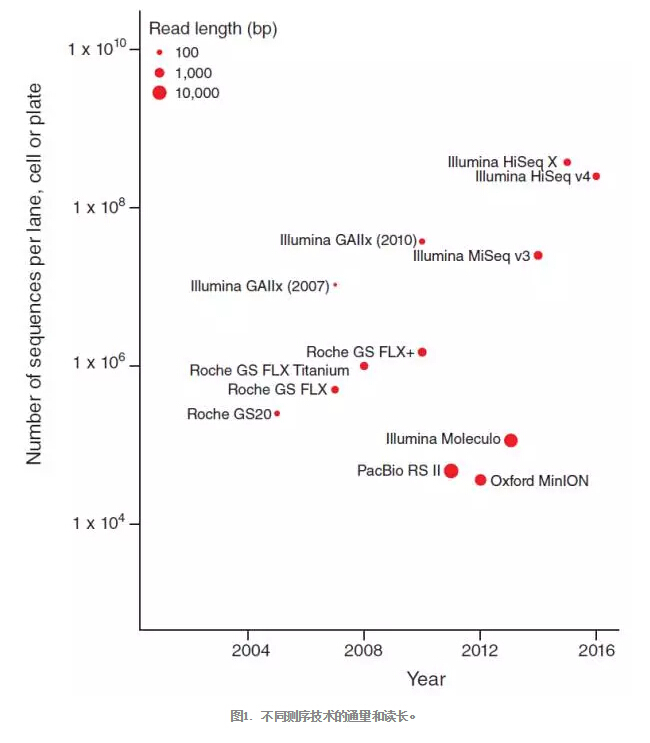
图1. 不同测序技术的通量和读长。
单分子测序技术
单分子测序技术是微生物组研究中新兴的测序技术。该技术中,首屈一指的当属PacBio的SMRT测序技术,该技术基于依靠拴系的DNA聚合酶和零模式波导来引导光能通过小体积的液体。PacBio提供的reads长度可以达到10-25 kbp,每个cell可以提供 ~300 Mbp数据。此外,该技术还可以检测特殊的或者修饰后的核酸碱基(不需要在合成过程中进行化学修饰),这是因为遇到特殊的碱基之后聚合酶会发生wobble。
另一种单分子测序平台为Oxford Nanopore,该平台主要依赖于被测核酸序列通过蛋白纳米孔时电导率的变化,通过监测电导率的变化进行测序,该平台同样可以检测被修饰的碱基,平均读长~1-2 kbp,有研究表明最长读长甚至达到>90 kbp,该平台最大的优点是可以利用无线技术传输数据并进行实时分析。
这些新兴技术的主要挑战是获得数百条长度为kbp甚至Mbp的高质量和大分子量的DNA,例如,PacBio测序平台需要微克级高分子量DNA(>40 kbp)用于文库构建。此外,这些技术的通量都相对比较低。但是对于序列组装方面的应用而言,这些单分子测序平台为微生物群落研究提供了一定的技术支撑。
微生物群落测序
DNA测序技术在发现、识别、鉴定微生物潜在功能方面发挥着重要作用,目前大部分的研究主要是多样性研究,即利用微生物基因组片段上一段扩增子序列进行细菌、真菌以及古菌的测序分析。此外,也有部分宏基因组方面的研究,利用该研究可以发现微生物群落中与某一特殊物种相关的功能基因。然而,大部分基因的功能目前还是未知的,这预示着对于环境微生物的研究依然任重而道远。
宏转录组
宏转录组技术是对某环境中所有微生物的mRNA进行测序分析,从而来探索某一时间或空间中特定微生物的基因表达情况。该技术有助于对宏基因组数据进行相关验证以及了解不同环境中微生物相对活性的变化情况。
2. 基于质谱技术的宏蛋白组学和宏代谢组学研究
尽管测序技术很大程度上加强了人们对于微生物系统发育以及功能基因组成的认识与理解,但是要想研究执行生命功能的蛋白质以及与其相关的代谢产物与环境等因素之间的关系,仅仅依靠这些数据还远远不够,因此需要借助质谱技术达到这一研究目的。在利用质谱技术进行微生物群落样本分析时,感兴趣的生物分子会被离子化,并根据质荷比分离开来,以达到检测的目的。质谱分析具有高灵敏度、高分辨率和高通量的特点。
自1984年electrospray ionization (ESI,电喷雾离子化技术)产生以来,利用质谱技术进行生物分子的研究日益剧增。电喷雾离子化技术的局限是需要在760 torr的气压下进行,但是质谱分析的气压要求在10-6 and 10-11 torr之间,两者间的气压差至少有9个数量级,如果离子化程序和质谱分析程序未能完美的契合,会导致大量离子流失。因此,在过去的20年,质谱研究主要聚焦在优化ion funnels和transmission quadrupole部分的接口设计,旨在解决衔接过程中减压问题,该部分的优化有利于降低离子流失并提高分析的敏感性。
然而,因为质谱结果的高动态范围以及样本中组成种类成千上万,所以利用质谱技术研究微生物群落生物分子存在一定的难点。为克服这一难点,要求质谱技术要有高分辨率、高准确性和高速度等特性。
在orbitrap(轨道阱)技术出现之前,Quadrupole( 四极杆质量分析质谱), ion trap(离子阱质谱), time-of-flight(飞行时间质谱)和ion cyclotron resonance (ICR,离子迥旋共振分析质谱)是主要的几种质谱技术。在过去的几十年中,orbitrap技术发展出了包括 linear ion trap (low resolution) 和orbitrap(high resolution)在内的具有高分辨率和高扫描率的技术。
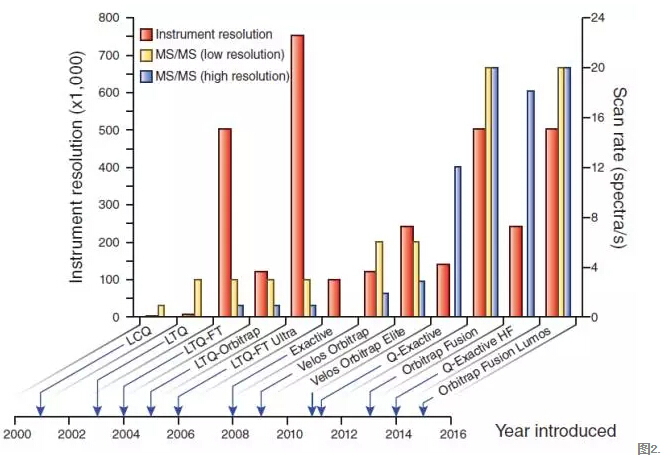
图2. 基于离子阱的质谱技术演变。
此外,分离技术的发展,例如一维和二维的液相色谱分离技术、气相离子迁移谱法都促进了微生物群落的研究。这些分离技术降低了样本检测前的复杂度,提高了检测样品中蛋白质和代谢物的覆盖率。
3.复杂环境微生物研究的局限和未来的方向
在过去几十年中,核酸测序技术以及质谱技术的发展使得研究复杂环境微生物群落结构成为可能。早在2000年,Banfield和其课题组成员首次结合高通量测序技术与质谱技术研究了酸性废矿水中多样性较低的微生物群落结构。随后,利用高通量测序技术、质谱技术或者两种技术结合进行微生物群落的研究日渐增长。随着技术的不断提高,人们期望对于微生物群落表型的认识能够更加深入。
图3. 微生物群落研究历程。
目前,对于微生物群落功能的认识与理解仍然是任重道远。当然,在该过程中依旧面临着很多挑战。其中最大的难题是生物信息学和计算方面瓶颈,包括非冗余基因集构建、微生物注释信息的完善和多组学结合的算法问题。面临的挑战还包括生物大分子的抽提;完整基因组数据的组装;利用质谱技术研究宏蛋白组、宏代谢组数据需要更快速、通量更高等。
其实,除了挑战,微生物群落研究的前景还是很宽广的。近来,白宫科学和技术政策办公室(OSTP)宣布启动新的国家微生物组计划(National Microbiome Initiative ,NMI),该计划旨在提高微生物研究的相关技术,推进对微生物群落的理解。人们坚信这项政策会更快的推进核酸测序技术以及质谱技术在微生物研究领域的应用,并获得更加深入的微生物群落的相关信息。此外,人们还期望,在这项政策的鼓舞下会诞生前所未有的新技术,例如,更高分辨率的肽段(或者代谢物)离子迁移分离手段或者新的可用于分子注释的数据库。
这些技术和计算的改进为破译微生物在其所处环境中扮演的角色以及其对其他生物,甚至对生态系统的健康与可持续性的影响在未来十年将发挥至关重要的作用。
如此深入了解微生物群落的表型将有助于更好地理解一些过程,例如气候变化、疾病对微生物功能的影响,使得我们能够更好的预知这些变化可能造成的影响,促进生态系统可持续发展和人类健康的策略。
